南京中医药大学第二临床医学院 210000
摘要 目的:通过分析GEO数据库中食管癌的芯片数据集,挖掘差异表达基因,并在TCGA数据库中验证,寻找食管癌的候选肿瘤标志物。方法:分析GEO数据库中食管癌的三个芯片数据集GSE17351、GSE75241、GSE89102,分别搜索差异表达基因,利用韦恩软件寻找核心基因。在TCGA数据库中验证核心基因,并且进行GO功能注释和KEGG代谢通路分析。最后对通过验证的核心基因进行生存分析。结果:GSE17351芯片数据集筛选出618个差异表达基因,其中下调基因265个,上调基因353个;GSE75241芯片数据集筛选出1652个差异表达基因,其中下调基因481个,上调基因1171个;GSE89102芯片数据集筛选出7939个差异表达基因,其中下调基因312个,上调基因7627个。利用韦恩软件,发现核心基因115个,上调基因94个,下调基因94个。在TCGA数据库中进行了验证、GO功能注释和KEGG代谢通路分析。对通过验证的核心基因进行生存分析,发现CDKN3、NUP155、RAD51AP1可以作为食管癌的候选肿瘤标志物。
关键词 食管癌、GEO、TCGA、肿瘤标志物
Abstract Objective: To analyze the differentially expressed genes by analyzing the microarray dataset of esophageal cancer in the GEO database, and to verify the candidate tumor markers of esophageal cancer in the TCGA database. METHODS: Three microarray data sets GSE17351, GSE75241 and GSE89102 of esophageal cancer in the GEO database were analyzed. The differentially expressed genes were searched and the core genes were searched using venn software. Core genes were verified in the TCGA database and GO functional annotation and KEGG pathway analysis were performed. Finally, survival analysis was performed on the verified core genes. RESULTS: The GSE17351 microarray dataset screened 618 differentially expressed genes, of which 265 were down-regulated and 353 were up-regulated. The GSE75241 microarray dataset screened out 1652 differentially expressed genes, of which 481 were down-regulated and 1171 were up-regulated; GSE89102 The data set screened 7939 differentially expressed genes, of which 312 were down-regulated and 7627 were up-regulated. Using venn software, 115 core genes were found, 94 genes were up-regulated, and 94 genes were down-regulated. Validation, GO functional annotation and KEGG pathway analysis were performed in the TCGA database. Survival analysis of the verified core genes revealed that CDKN3, NUP155, and RAD51AP1 can be used as candidate tumor markers for esophageal cancer.
Key words esophageal cancer, GEO, TCGA, tumor markers
食管癌(esophageal cancer)又叫食道癌,是一种发生于食管上皮的恶性肿瘤,是临床上最为常见的消化道恶性肿瘤疾病之一[1]。食管癌在世界恶性肿瘤排名第八,其死亡率居所有癌症第六[2]。最近几年,全球食管癌的发病率呈上升趋势。据世界卫生组织2012年的报告显示,全球新增食管癌患者人数已经达到455800。值得一提的是,中国食管癌的新发病例和死亡率均居世界第一。食管癌的发病年龄一般在40岁以上,男性发病率高于女性,目前已经成为严重威胁人类健康的常见病种[3]。食管癌患者的早期临床症状不明显,当发现临床不适症状时,病情已经进入中晚期,从而失去了外科治疗、放疗和化疗等最佳治疗时机。因此,只有做到早诊早治才能降低食管癌死亡率。本文通过GEO数据库和TCGA数据库联合分析,筛选出食管癌差异表达基因,同时计算差异表达基因的生存曲线,为食管癌的早期诊断提供新的候选肿瘤标志物。
1 材料与方法
1.1 GEO数据库芯片数据分析
本研究首先在NCBI GEO(https://www.ncbi.nlm.nih.gov/geo/)数据库检索关键词esophageal+cancer+Homo sapiens,共检索到2147个芯片数据集。然后选择GSE17351、GSE75241和GSE89102作为食管癌的分析数据,其中GSE17351包含5例食管癌组织样本和5例正常组织样本,GSE75241包含15例食管癌组织样本和15例正常组织样本,GSE89102包含5例食管癌组织样本和5例正常组织样本。
1.2 差异表达基因的筛选
点击GSE芯片数据集编号,进入芯片数据集,使用GEO数据库自带的差异表达基因分析工具GEO2R分析数据集的差异表达基因。分析数据前首先需要点击Define groups进行数据分组:癌症组与正常组,然后点击下方方框中的TOP250进行差异表达基因分析。点击save all results可以得到所有基因的差异表达结果,然后根据筛选条件筛选差异表达基因。本文的差异表达基因筛选条件为p.adj<0.05且|logFC|>1。
1.3 核心基因的筛选
取上述三个芯片数据集所得的差异表达基因的交集作为食管癌的核心基因,使用韦恩图绘制软件绘制三个芯片数据集所得的差异表达基因的韦恩图。
1.4 核心基因的验证与GO功能注释、KEGG代谢通路分析
使用TCGA数据库在线分析网站UALCAN[4](http://ualcan.path.uab.edu/)分析核心基因在食管癌中的表达。对通过验证的核心基因进行GO功能注释和KEGG代谢通路分析。
1.5 生存曲线分析
使用TCGA数据库在线分析网站UALCAN(http://ualcan.path.uab.edu/)分析通过验证的核心基因对食管癌生存曲线的影响。
2 结果
2.1 GSE17351芯片数据集差异表达基因分析结果
点击GSE17351编号,进入芯片数据集,使用GEO数据库自带的差异表达基因分析工具GEO2R分析数据集的差异表达基因。分析数据前首先点击Define groups进行数据分组:癌症组与正常组,然后点击下方方框中的TOP250进行差异表达基因分析。点击save all results得到所有基因的差异表达结果,然后根据筛选条件p.adj<0.05且|logFC|>1筛选差异表达基因。共筛选出618个差异表达基因,其中下调基因265个,上调基因353个。本数据集的差异表达基因火山图如图1所示。
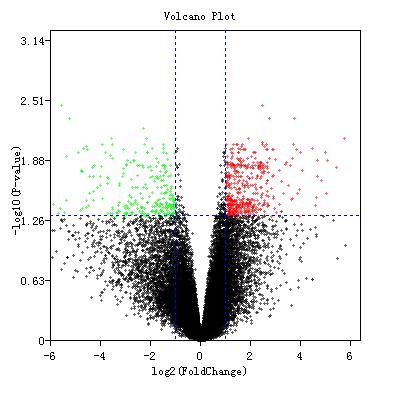
图1 GSE17351的差异表达基因火山图
Figure 1 The volcanic map of differentially expressed genes of GSE17351
2.2 GSE75241芯片数据集差异表达基因分析结果
点击GSE75241编号,进入芯片数据集,使用GEO数据库自带的差异表达基因分析工具GEO2R分析数据集的差异表达基因。分析数据前首先点击Define groups进行数据分组:癌症组与正常组,然后点击下方方框中的TOP250进行差异表达基因分析。点击save all results得到所有基因的差异表达结果,然后根据筛选条件p.adj<0.05且|logFC|>1筛选差异表达基因。共筛选出1652个差异表达基因,其中下调基因481个,上调基因1171个。本数据集的差异表达基因火山图如图2所示。
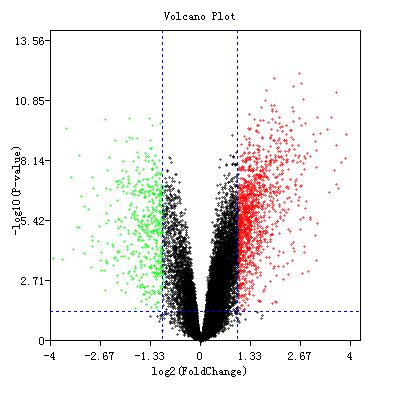
图2 GSE75241的差异表达基因火山图
Figure 2 The volcanic map of differentially expressed genes of GSE75241
2.3 GSE89102芯片数据集差异表达基因分析结果
点击GSE89102编号,进入芯片数据集,使用GEO数据库自带的差异表达基因分析工具GEO2R分析数据集的差异表达基因。分析数据前首先点击Define groups进行数据分组:癌症组与正常组,然后点击下方方框中的TOP250进行差异表达基因分析。点击save all results得到所有基因的差异表达结果,然后根据筛选条件p.adj<0.05且|logFC|>1筛选差异表达基因。共筛选出7939个差异表达基因,其中下调基因312个,上调基因7627个。本数据集的差异表达基因火山图如图3所示。

图3 GSE89102的差异表达基因火山图
Figure 3 The volcanic map of differentially expressed genes of GSE89102
2.4 核心基因的筛选
使用venn软件对上述三个芯片数据集所得的差异表达基因进行韦恩分析,发现GSE17351、GSE75241和GSE89102三个芯片数据集共有115个共同表达的差异表达基因,其中下调基因21个,上调基因94个。这些共同表达的差异表达基因可以作为食管癌的核心基因,GSE17351、GSE75241和GSE89102三个芯片数据集的韦恩图如图4所示。核心基因的上下调情况如表1所示。

图4三个芯片数据集差异表达基因的韦恩图
Figure 4 Venn diagram of differentially expressed genes in three GEO datasets
表1 核心基因的上调基因和下调基因列表
Table 1 List of up-regulated genes and down-regulated genes of core genes
| Type | Number | Gene name |
| Down-Regulated | 21 | TMPRSS11B HPGD SPINK5 SH3BGRL2 GBP6 ABLIM3 BBOX1 KAT2B NUCB2 SHROOM3 DDAH1 CYP4F12 SIM2 CYP11A1 ANKRD35 CAPNS KANK1 CBR3 PAQR8 FCHO2 SLC6A1 |
| Up-Regulated | 94 | DNMT1 ADAM12 CENPQ LAMC2 GMPS UHRF1 MAD2L1 MND1 MCM10 TTK DSCC1 CENPI KIF20A TPX2 MMP11 KNTC1 RFC3 AURKA UBE2T NUF2 DTL CDCA3 CKAP2L RAD51AP1 VRK1 NUP155 CDC6 SPC25 ARPC1B CDH3 FZD6 ANP32E CKAP5 PRKDC PTDSS1 CCT5 CHML RUVBL1 HELLS CENPK TRIP13 RSRC1 ATP2C1 HEATR1 HEATR1 LAMB3 PARP1 CTSC GMNN GORAB CD276 IGF2BP2 ABCC4 HLTF TMEM39A MMP10 ITGA6 ATP1B3 WDR6 MMP13 SLC25A32 ITGB4 E2F6 TMEM97 ATP6V1C1 IPO9 LRRC8D RPS6KA1 GCA RBMS3 TFPI ATP2B1 OASL NAPEPLD ZAK ENAH CDCA2 RAD54B MCM5 HOXC10 STMN1 KIF23 DEPDC1 CTHRC1 CDKN3 ATAD2 TFRC CDC25B TOP2A KIF18A KIF14 |
2.5 核心基因在TCGA数据库中的验证
打开TCGA数据库在线分析网站UALCAN(http://ualcan.path.uab.edu/),在食管癌中检索核心基因。TCGA数据库中食管癌样本有189例,食管正常组织样本11例。结果显示下调的21个核心基因有4个通过验证,上调的94个核心基因有86个通过验证。通过验证的上下调核心基因列表如表2所示。
表2 通过验证的上下调核心基因列表
Table 2 List of up-regulated genes and down-regulated genes of verified core genes
| Type | Number | Gene name |
| Down-Regulated | 21 | HPGD KAT2B NUCB2 DDAH1 |
| Up-Regulated | 94 | DNMT1 ADAM12 CENPQ LAMC2 GMPS UHRF1 MAD2L1 MND1 MCM10 TTK DSCC1 CENPI KIF20A TPX2 MMP11 KNTC1 RFC3 AURKA UBE2T NUF2 DTL CDCA3 CKAP2L RAD51AP1 VRK1 NUP155 CDC6 SPC25 ARPC1B CDH3 FZD6 ANP32E CKAP5 PRKDC PTDSS1 CCT5 CHML RUVBL1 HELLS CENPK TRIP13 RSRC1 ATP2C1 HEATR1 HEATR1 LAMB3 PARP1 CTSC GMNN GORAB CD276 IGF2BP2 ABCC4 HLTF TMEM39A MMP10 ITGA6 ATP1B3 WDR6 MMP13 SLC25A32 ITGB4 E2F6 TMEM97 ATP6V1C1 IPO9 LRRC8D RPS6KA1 CDCA2 RAD54B MCM5 HOXC10 STMN1 KIF23 DEPDC1 CTHRC1 CDKN3 ATAD2 TFRC CDC25B TOP2A KIF18A KIF14 |
2.6通过验证的核心基因的GO功能注释、KEGG代谢通路分析
为了研究通过验证的核心基因的生物学功能和参与的代谢途径,我们使用R语言中的clusterProfiler[5]软件包对上述基因分别进行GO功能注释和KEGG代谢通路分析。GO功能注释结果显示,通过验证的核心基因在生物进程(BP)中主要表现在:姐妹染色单体分离、核染色体分离、细胞器裂变、有丝分裂核分裂;在细胞组份(CC)中主要表现在:染色体区域、纺锤、着丝点、有丝分裂纺锤体;在在分子功能(MF)主要表现在:ATP酶活性、DNA依赖性ATP酶活性、活性离子跨膜转运蛋白活性、ATPase偶联离子跨膜转运蛋白活性(图5)。KEGG代谢通路分析结果显示食管癌的核心基因主要集中在细胞周期、ECM-受体相互作用、DNA复制、细胞粘附分子(CAMs)、mTOR信号通路等代谢通路(图6)。
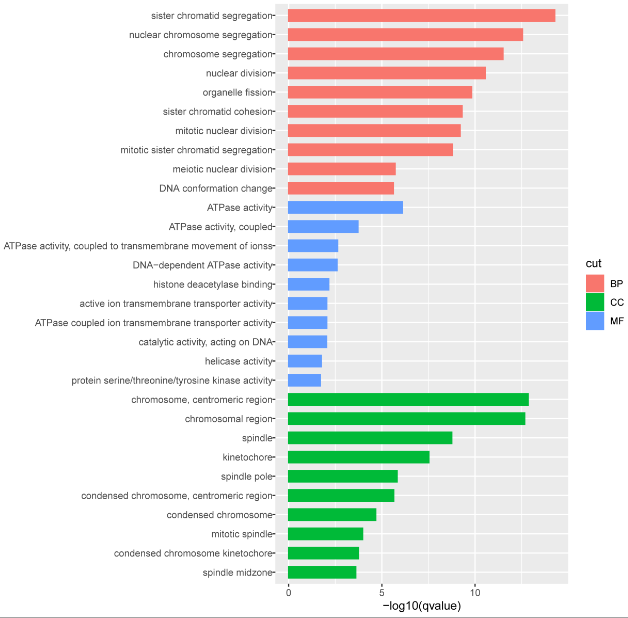
图5 通过验证的核心基因的GO分析
Figure 5 GO analysis of verified core genes
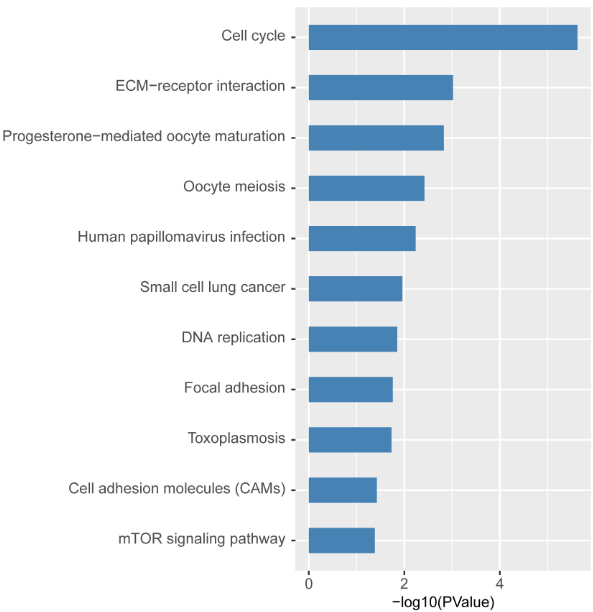
图6 通过验证的核心基因的KEGG分析
Figure 6 KEGG analysis of verified core genes
2.7 通过验证的核心基因的生存曲线
在TCGA数据中共有189例食管癌样本芯片数据,其中184例样本有生存数据。我们对通过验证的核心基因进行生存分析,发现46例CDKN3、NUP155、RAD51AP1基因表达升高患者的生存时间明显低于138例CDKN3、NUP155、RAD51AP1基因表达降低患者的生存时间,说明CDKN3、NUP155、RAD51AP1对食管癌具有促进作用(图7)。
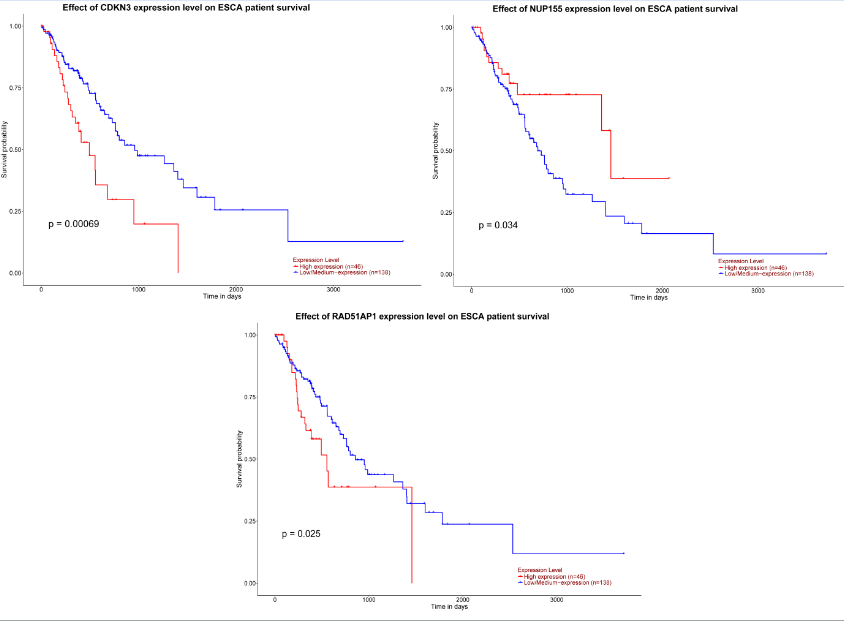
图7 通过验证的核心基因的生存分析
Figure 7 Survival analysis of verified core genes
3 讨论
食管癌(esophageal cancer)是一种发生于食管上皮的恶性肿瘤,是临床上最为常见的消化道恶性肿瘤疾病之一,主要包括食管鳞癌和食管腺癌两种亚型。最近几年,全球食管癌的发病率呈上升趋势。食管癌的发病年龄一般在40岁以上,男性发病率高于女性。食管癌的发病呈现出区域性特征,因而致病因素非常复杂,目前普遍认为食管癌是多因素共同作用导致的。食管癌患者的早期临床症状不明显,没有引起患者的重视,当产生临床不适症状时,病情已经进入中晚期,从而失去了外科治疗、放疗和化疗等最佳治疗时机。因此,筛选食管癌的肿瘤标志物对食管癌的早诊早治至关重要。
[1] Jemal A, Bray F, Center M M, et al. Global cancer statistics[J]. CA: a cancer journal for clinicians, 2011, 61(2): 69-90.
[2] Stoner G D, Gupta A. Etiology and chemoprevention of esophageal squamous cell carcinoma[J]. Carcinogenesis, 2001, 22(11): 1737-1746.
[3] 赵敦梅. 食管鳞癌中联合 miRNA 和 DNA 甲基化相关基因的诊断模型建立及候选基因 TLX2 在食管鳞癌发生发展中的研究[D]. 苏州大学, 2017.
[4] Chandrashekar D S, Bashel B, Balasubramanya S A H, et al. UALCAN: a portal for facilitating tumor subgroup gene expression and survival analyses[J]. Neoplasia, 2017, 19(8): 649-658.
[5] Yu G, Wang L G, Han Y, et al. clusterProfiler: an R package for comparing biological themes among gene clusters[J]. Omics: a journal of integrative biology, 2012, 16(5): 284-287.